1. 一房室口服
选择口服给药、符合单室模型的药物来画图,因为这种药物较为常见,公式也很简单,并将2个模型的血药浓度画在同一张图上,便于比较。
1.1 单剂量给药
首先绘制单剂量给药,对于model 1,我们设置给药剂量,生物利用度,表观分布容积,一级吸收速率常数,一级消除速率常数;对于model 2,我们设置给药剂量,生物利用度,表观分布容积,一级吸收速率常数,一级消除速率常数。
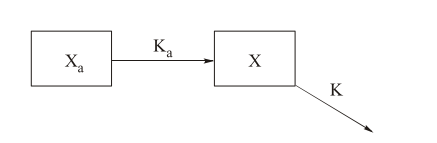
隔室模型示意图如下:
model 1的血药浓度表达式为:
model 2的血药浓度表达式为:
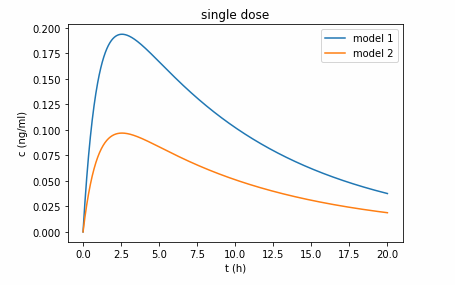
有了血药浓度公式,下面就可以画图了:
import numpy as np
import matplotlib.pyplot as plt
e=np.e
plt.figure(figsize=(6,4))
t= np.linspace(0, 20, 1000)
plt.plot(t,5/(20*(1-0.1))*(e**(-0.1*t)-e**(-1*t)),label='model 1')
plt.legend()
plt.plot(t,5/(40*(1-0.1))*(e**(-0.1*t)-e**(-1*t)),label='model 2')
plt.legend()
plt.title('single dose')
plt.xlabel('t (h)')
plt.ylabel('c (ng/ml)')
plt.show()
1.2 多剂量给药
单剂量搞定,下面考虑多剂量给药的图形。对于多剂量给药,我们一般需要使血药浓度达到稳态。
以model1为例,首先求算其表达式。设置给药间隔,给药次数。在第1个给药周期内,其血药浓度为第1次给药后的浓度;在第2个给药周期内,其血药浓度为第1次给药后5~10 h内的浓度加上第2次给药后0~5 h内的浓度;以此类推。显然,多剂量给药,其体内药量是一个分段函数:
1.2.1 直接画
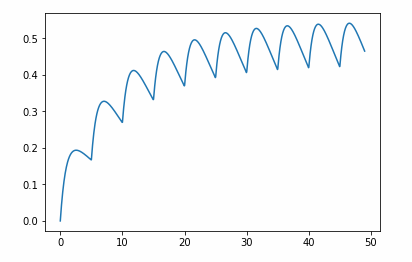
分段函数图像分段画,代码如下:
import numpy as np
import matplotlib.pyplot as plt
e=np.e
t= np.linspace(0,49,1000)
#intervals
i1 = [1 if (i>=0 and i<=5) else 0 for i in t]
i2 = [1 if (i>=5 and i<=10) else 0 for i in t]
i3 = [1 if (i>=10 and i<=15) else 0 for i in t]
i4 = [1 if (i>=15 and i<=20) else 0 for i in t]
i5 = [1 if (i>=20 and i<=25) else 0 for i in t]
i6 = [1 if (i>=25 and i<=30) else 0 for i in t]
i7 = [1 if (i>=30 and i<=35) else 0 for i in t]
i8 = [1 if (i>=35 and i<=40) else 0 for i in t]
i9 = [1 if (i>=40 and i<=45) else 0 for i in t]
i10 = [1 if (i>=45 and i<=49) else 0 for i in t]
def c(t):
return 5/(20*(1-0.1))*(e**(-0.1*t)-e**(-1*t))
y=c(t)*i1+\
(c(t)+c(t-5))*i2+\
(c(t)+c(t-5)+c(t-10))*i3+\
(c(t)+c(t-5)+c(t-10)+c(t-15))*i4+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20))*i5+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20)+c(t-25))*i6+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20)+c(t-25)+c(t-30))*i7+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20)+c(t-25)+c(t-30)+c(t-35))*i8+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20)+c(t-25)+c(t-30)+c(t-35)+c(t-40))*i9+\
(c(t)+c(t-5)+c(t-10)+c(t-15)+c(t-20)+c(t-25)+c(t-30)+c(t-35)+c(t-40)+c(t-45))*i10
plt.plot(t,y)
plt.show()
画出model1给药次数为10次,给药间隔为5h时的药时曲线:
1.2.2 根据多剂量函数画
由于分段函数需要分段表示,必须要用语句划分区间,并且每一段的表达式都要写出来,导致上述代码比较长。在药代动力学教材中,通过引入多剂量函数()这个概念,可利用和把第次给药的体内药量的通式表示出来:
则第次给药后,血药浓度为:
根据此公式画图:
import numpy as np
import matplotlib.pyplot as plt
e=np.e
t= np.linspace(0,49,1000)
i1 = [1 if (i>=0 and i<=5) else 0 for i in t]
i2 = [1 if (i>=5 and i<=10) else 0 for i in t]
i3 = [1 if (i>=10 and i<=15) else 0 for i in t]
i4 = [1 if (i>=15 and i<=20) else 0 for i in t]
i5 = [1 if (i>=20 and i<=25) else 0 for i in t]
i6 = [1 if (i>=25 and i<=30) else 0 for i in t]
i7 = [1 if (i>=30 and i<=35) else 0 for i in t]
i8 = [1 if (i>=35 and i<=40) else 0 for i in t]
i9 = [1 if (i>=40 and i<=45) else 0 for i in t]
i10 = [1 if (i>=45 and i<=49) else 0 for i in t]
def c(t,n):
return 5/(20*(1-0.1))*((1-e**(-0.1*5*n))/(1-e**(-5*0.1))*e**(-0.1*t)-(1-e**(-1*5*n))/(1-e**(-1*5))*e**(-1*t))
y=c(t,1)*i1+c(t-5,2)*i2+c(t-10,3)*i3+c(t-15,4)*i4+c(t-20,5)*i5+c(t-25,6)*i6+c(t-30,7)*i7+c(t-35,8)*i8+c(t-40,9)*i9+c(t-45,10)*i10
plt.plot(t,y)
plt.show()
1.2.3 给药次数为
与上一段代码比起来,这段代码在表示y的时候简洁了许多,但仍然需要划分区间,这就导致在改变给药次数后,画图需要改动代码,并且比较大时,代码也比较长。那么如在让我们设置任意的时,图形都可以方便地画出来呢?
之前我们是根据表达式直接画图(虽然Python实现起来是通过生成很多散点),而表达式一旦是分段函数的话,画起来就很麻烦,必须分段(因此可以看到代码中有很长的一段是在分割区间)。现在可以转换一下思路,不通过表达式画图,而是通过表达式生成散点,再通过散点作图。利用分段函数产生点比利用分段函数画图方便多了,尤其是C(n,t)的通式已经给出了,只需一个简单的循环语句就能生成每一次给药后的浓度点,再通过Python的列表(list)或数组(array)的append()方法,把不同时间段的浓度点连接起来,就是我们所需要的(全程的浓度),根据时间散点和浓度散点,就可以把图画出来了。并且给药次数可以任意设置。
给药次数设为50次:
import numpy as np
import matplotlib.pyplot as plt
def c(t,n):
return 5/(20*(1-0.1))*((1-e**(-0.1*5*n))/(1-e**(-5*0.1))*e**(-0.1*t)-(1-e**(-1*5*n))/(1-e**(-1*5))*e**(-1*t))
e=np.e
t1= np.linspace(0,4,100)
y1=c(t1,1)
t=t1
y=y1
for N in range(2,50):
tN=t1+(N-1)*5
yN=c(t1,N)
t=np.append(t,tN)
y=np.append(y,yN)
plt.plot(t,y)
plt.show()
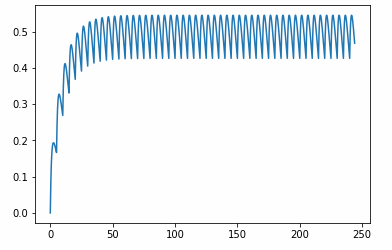
1.3 最终画图结果
由于开头说比较model 1和model 2,要把2条曲线画在一起。所以最后再画上model 2的药时曲线,添上标题、图例、横纵坐标,画图完成。
import numpy as np
import matplotlib.pyplot as plt
plt.figure(figsize=(8,6))
def c1(t,n):
return 5/(20*(1-0.1))*((1-e**(-0.1*5*n))/(1-e**(-5*0.1))*e**(-0.1*t)-(1-e**(-1*5*n))/(1-e**(-1*5))*e**(-1*t))
def c2(t,n):
return 5/(20*(1-0.2))*((1-e**(-0.2*5*n))/(1-e**(-5*0.2))*e**(-0.2*t)-(1-e**(-1*5*n))/(1-e**(-1*5))*e**(-1*t))
e=np.e
t0= np.linspace(0,4,100)
t=t0
y1=c1(t0,1)
y2=c2(t0,1)
for N in range(2,20):
tN=t0+(N-1)*5
yN1=c1(t0,N)
yN2=c2(t0,N)
t=np.append(t,tN)
y1=np.append(y1,yN1)
y2=np.append(y2,yN2)
plt.plot(t,y1,label='model 1')
plt.plot(t,y2,label='model 2')
plt.legend()
plt.title('multiple dose')
plt.xlabel('t (h)')
plt.ylabel('c (ng/ml)')
plt.show()
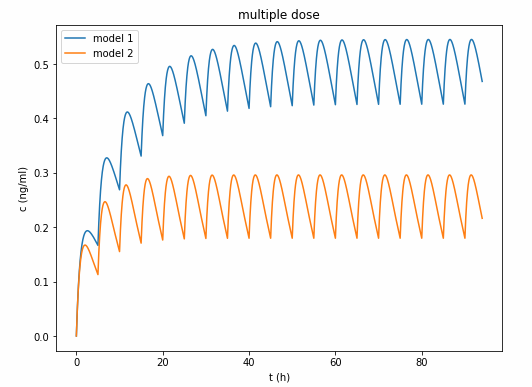
2. 母药和代谢物
有些药物本身不具有药理活性,进入体内后经代谢产生活性物质,也有一些药物,其自身与代谢物都具有药理活性,对于此类药物,我们需要关注它们的代谢物的浓度。
利用隔室模型进行研究时,步骤是根据隔室模型写出微分方程组,求出表达式,画图。对于常见的一室、二室、三室模型,药代动力学课本上都给出了血药浓度公式,但是有了代谢物之后,就不是应用上述模型,换句话说,没有课本上现成的公式可以套用了。
那么如何画图呢?我们一般会想到2种方法:
- 根据微分方程组解出方程,通过方程画图;
- 直接根据微分方程画图。
对于以上2种方法,可以综合使用。
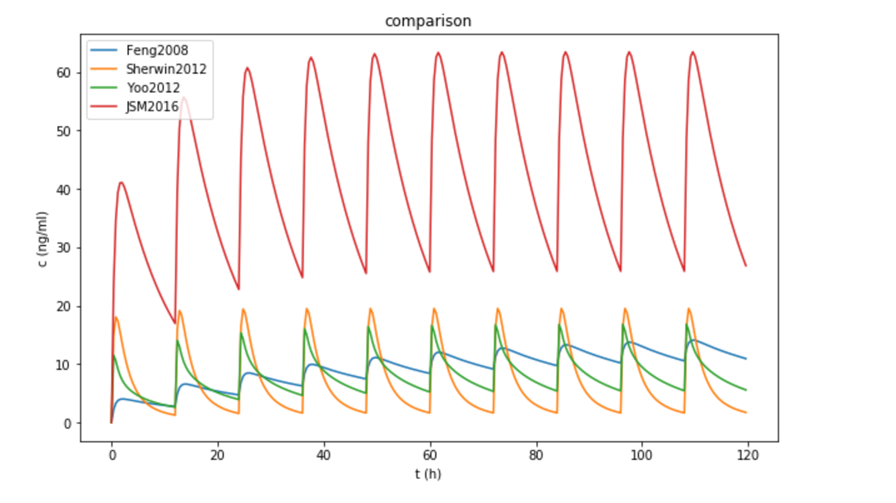
下面以抗精神分裂药利培酮(Risperidone)为例,画出其母药、代谢物以及二者之和的浓度曲线。利培酮在体内代谢为9-羟利培酮,它们都具有药理活性,近年来国内外有不少文献使用群体建模方法对该药物进行了研究。本文画图所使用的的PK参数来自如下文献:
-
model 1:Feng2008
-
model 2:Sherwin2012
-
model 3:Yoo2012
-
model 4:JSM2016
2.1 model1单剂量
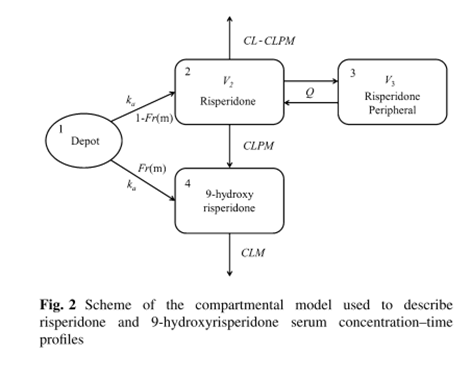
先解决绘制model1的问题。文献中给出了隔室模型的示意图:
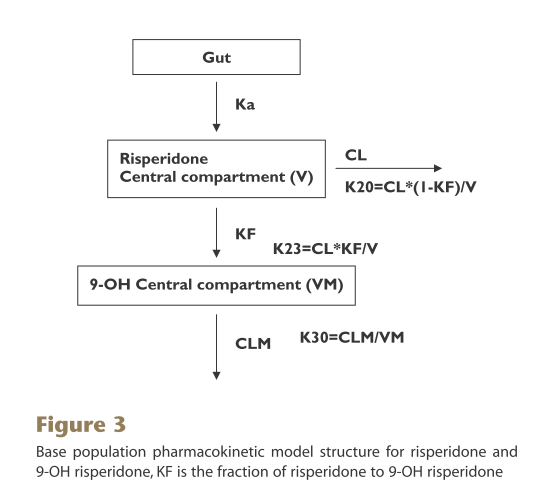
根据上图,写出微分方程组:
其中(1)代表吸收部位药量,(2)代表利培酮药量,(3)代表9-羟利培酮药量。
从文献中找到PK参数的值:,,,,,,我们设置给药剂量为2mg。
将参数带入微分方程组,可以算出方程(1)表达式:
将参数和式(4)带入方程(2),方程可写为:
可求出表达式:
将参数和式(6)带入方程(3),方程可写为:
方程(7)暂时先不解。
有了以上的式子,可以着手用Python画图了。scipy这个科学计算库可以根据微分方程表达式直接画出图形而不必解方程。上面之所以解出了(1)和(2)的表达式,是因为(2)和(3)的方程里分别含有和。
# Feng2008 单剂量
import numpy as np
from scipy.integrate import odeint
import matplotlib.pyplot as plt
e=np.e
def diff_x2(x,t):
return 1.7*2*e**(-1.7*t)-0.148*x
def diff_x3(x,t):
return 0.088*2.19*(e**(-0.148*t)-e**(-1.7*t))-0.02*x
t=np.arange(0,100,0.01)
x2=odeint(diff_x2,0,t)
x3=odeint(diff_x3,0,t)
c2=x2/444*1000
c3=x3/444*1000
c_risperidone=c2+c3
plt.plot(t,c2,label='RISP')
plt.plot(t,c3,label='9-OH')
plt.plot(t,c_risperidone,label='RISP+9-OH')
plt.legend()
plt.title('Feng2008 single dose')
plt.xlabel('t (h)')
plt.ylabel('c (ng/ml)')
plt.show()
这段代码画出了利培酮(RISP)、9-羟利培酮(9-OH)以及二者浓度之和(RISP+9-OH):
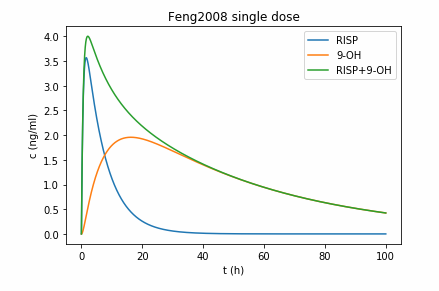
图画出来了,还有一些我们值得思考的地方:
第一,如上所述,解微分方程时,我们是笔算的,能否通过Python来求解微分方程?
第二,我们写出的其实一个微分方程组,但实际画图时是把它们当做独立的微分方程来画图,能否直接通过线性(微分)方程组画图?
对于问题一,答案是Python是可以解微分方程的。科学计算库sympy是一种方法,解式(1):
import sympy as sy
def differential_equation(x,f):
return sy.diff(f(x),x,1)+1.7*f(x)
x=sy.symbols('t')
f=sy.Function('Ca')
print(sy.dsolve(differential_equation(x,f),f(x)))
解得表达式如下:
Eq(Ca(t), C1exp(-1.7t))
这个结果与笔算的结果一致,是正确的。
解式(2) :
import numpy as np
import sympy as sy
e=np.e
def differential_equation(x,f):
return sy.diff(f(x),x,1)-3.4*e**(-1.7*x)+0.148*f(x)
x=sy.symbols('x')
f=sy.Function('f')
print(sy.dsolve(differential_equation(x,f),f(x)))
结果如下:
Eq(f(x), (C1 - 2.19072164948454exp(-1.552x))exp(-0.148x))
这个结果是错误的,改变一下写法:
# -- coding:utf-8 --
from sympy import *
#用sympy符号运算解方程
x=symbols('x',real = True) # real 保证全是实数,自变量
y=symbols('y',function = True) # 全部为函数变量
eq=y(x).diff(x,1)-3.4*exp(-1.7*x)+0.148*y(x)
print(dsolve(Eq(eq,0),y(x)))
结果依然如此。错误的原因我还没有找到,此问题悬而未决。对于问题二,目前我也没找到相关的方法,也有待解决。
2.2 model 1多剂量
画出了model 1的单剂量给药的血药浓度图,多剂量该如何画呢?参考画一室模型多剂量给药的方法,先生成散点,由于没有借助通式,代码非常长。
# Feng2008 多剂量
import numpy as np
from scipy.integrate import odeint
import matplotlib.pyplot as plt
plt.figure(figsize=(8,4))
e=np.e
def diff_x2(x,t):
return 1.7*2*e**(-1.7*t)-0.148*x
def diff_x3(x,t):
return 0.088*2.19*(e**(-0.148*t)-e**(-1.7*t))-0.02*x
t1=np.arange(0,120,0.4)
x2_1=odeint(diff_x2,0,t1)
x2_11=x2_1[range(0,30)]
x2_12=x2_1[range(30,60)]
x2_13=x2_1[range(60,90)]
x2_14=x2_1[range(90,120)]
x2_15=x2_1[range(120,150)]
x2_16=x2_1[range(150,180)]
x2_17=x2_1[range(180,210)]
x2_18=x2_1[range(210,240)]
x2_19=x2_1[range(240,270)]
x2_110=x2_1[range(270,300)]
x3_1=odeint(diff_x3,0,t1)
x3_11=x3_1[range(0,30)]
x3_12=x3_1[range(30,60)]
x3_13=x3_1[range(60,90)]
x3_14=x3_1[range(90,120)]
x3_15=x3_1[range(120,150)]
x3_16=x3_1[range(150,180)]
x3_17=x3_1[range(180,210)]
x3_18=x3_1[range(210,240)]
x3_19=x3_1[range(240,270)]
x3_110=x3_1[range(270,300)]
t2=np.arange(12,120,0.4)-12
x2_2=odeint(diff_x2,0,t2)
x2_22=x2_2[range(0,30)]
x2_23=x2_2[range(30,60)]
x2_24=x2_2[range(60,90)]
x2_25=x2_2[range(90,120)]
x2_26=x2_2[range(120,150)]
x2_27=x2_2[range(150,180)]
x2_28=x2_2[range(180,210)]
x2_29=x2_2[range(210,240)]
x2_210=x2_2[range(240,270)]
x3_2=odeint(diff_x3,0,t2)
x3_22=x3_2[range(0,30)]
x3_23=x3_2[range(30,60)]
x3_24=x3_2[range(60,90)]
x3_25=x3_2[range(90,120)]
x3_26=x3_2[range(120,150)]
x3_27=x3_2[range(150,180)]
x3_28=x3_2[range(180,210)]
x3_29=x3_2[range(210,240)]
x3_210=x3_2[range(240,270)]
t3=np.arange(24,120,0.4)-24
x2_3=odeint(diff_x2,0,t3)
x2_33=x2_3[range(0,30)]
x2_34=x2_3[range(30,60)]
x2_35=x2_3[range(60,90)]
x2_36=x2_3[range(90,120)]
x2_37=x2_3[range(120,150)]
x2_38=x2_3[range(150,180)]
x2_39=x2_3[range(180,210)]
x2_310=x2_3[range(210,240)]
x3_3=odeint(diff_x3,0,t3)
x3_33=x3_3[range(0,30)]
x3_34=x3_3[range(30,60)]
x3_35=x3_3[range(60,90)]
x3_36=x3_3[range(90,120)]
x3_37=x3_3[range(120,150)]
x3_38=x3_3[range(150,180)]
x3_39=x3_3[range(180,210)]
x3_310=x3_3[range(210,240)]
t4=np.arange(36,120,0.4)-36
x2_4=odeint(diff_x2,0,t4)
x2_44=x2_4[(range(0,30))]
x2_45=x2_4[range(30,60)]
x2_46=x2_4[range(60,90)]
x2_47=x2_4[range(90,120)]
x2_48=x2_4[range(120,150)]
x2_49=x2_4[range(150,180)]
x2_410=x2_4[range(180,210)]
x3_4=odeint(diff_x3,0,t4)
x3_44=x3_4[(range(0,30))]
x3_45=x3_4[range(30,60)]
x3_46=x3_4[range(60,90)]
x3_47=x3_4[range(90,120)]
x3_48=x3_4[range(120,150)]
x3_49=x3_4[range(150,180)]
x3_410=x3_4[range(180,210)]
t5=np.arange(48,120,0.4)-48
x2_5=odeint(diff_x2,0,t5)
x2_55=x2_5[range(0,30)]
x2_56=x2_5[range(30,60)]
x2_57=x2_5[range(60,90)]
x2_58=x2_5[range(90,120)]
x2_59=x2_5[range(120,150)]
x2_510=x2_5[range(150,180)]
x3_5=odeint(diff_x3,0,t5)
x3_55=x3_5[range(0,30)]
x3_56=x3_5[range(30,60)]
x3_57=x3_5[range(60,90)]
x3_58=x3_5[range(90,120)]
x3_59=x3_5[range(120,150)]
x3_510=x3_5[range(150,180)]
t6=np.arange(60,120,0.4)-60
x2_6=odeint(diff_x2,0,t6)
x2_66=x2_6[range(0,30)]
x2_67=x2_6[range(30,60)]
x2_68=x2_6[range(60,90)]
x2_69=x2_6[range(90,120)]
x2_610=x2_6[range(120,150)]
x3_6=odeint(diff_x3,0,t6)
x3_66=x3_6[range(0,30)]
x3_67=x3_6[range(30,60)]
x3_68=x3_6[range(60,90)]
x3_69=x3_6[range(90,120)]
x3_610=x3_6[range(120,150)]
t7=np.arange(72,120,0.4)-72
x2_7=odeint(diff_x2,0,t7)
x2_77=x2_7[range(0,30)]
x2_78=x2_7[range(30,60)]
x2_79=x2_7[range(60,90)]
x2_710=x2_7[range(90,120)]
x3_7=odeint(diff_x3,0,t7)
x3_77=x3_7[range(0,30)]
x3_78=x3_7[range(30,60)]
x3_79=x3_7[range(60,90)]
x3_710=x3_7[range(90,120)]
t8=np.arange(84,120,0.4)-84
x2_8=odeint(diff_x2,0,t8)
x2_88=x2_8[range(0,30)]
x2_89=x2_8[range(30,60)]
x2_810=x2_8[range(60,90)]
x3_8=odeint(diff_x3,0,t8)
x3_88=x3_8[range(0,30)]
x3_89=x3_8[range(30,60)]
x3_810=x3_8[range(60,90)]
t9=np.arange(96,120,0.4)-96
x2_9=odeint(diff_x2,0,t9)
x2_99=x2_9[range(0,30)]
x2_910=x2_9[range(30,60)]
x3_9=odeint(diff_x3,0,t9)
x3_99=x3_9[range(0,30)]
x3_910=x3_9[range(30,60)]
t10=np.arange(108,120,0.4)-108
x2_10=odeint(diff_x2,0,t7)
x2_1010=x2_10[range(0,30)]
x3_10=odeint(diff_x3,0,t10)
x3_1010=x3_10[range(0,30)]
x21=x2_11
x22=x2_12+x2_22
x23=x2_13+x2_23+x2_33
x24=x2_14+x2_24+x2_34+x2_44
x25=x2_15+x2_25+x2_35+x2_45+x2_55
x26=x2_16+x2_26+x2_36+x2_46+x2_56+x2_66
x27=x2_17+x2_27+x2_37+x2_47+x2_57+x2_67+x2_77
x28=x2_18+x2_28+x2_38+x2_48+x2_58+x2_68+x2_78+x2_88
x29=x2_19+x2_29+x2_39+x2_49+x2_59+x2_69+x2_79+x2_89+x2_99
x210=x2_110+x2_210+x2_310+x2_410+x2_510+x2_610+x2_710+x2_810+x2_910+x2_1010
x2=np.concatenate((x21,x22,x23,x24,x25,x26,x27,x28,x29,x210))
x31=x3_11
x32=x3_12+x3_22
x33=x3_13+x3_23+x3_33
x34=x3_14+x3_24+x3_34+x3_44
x35=x3_15+x3_25+x3_35+x3_45+x3_55
x36=x3_16+x3_26+x3_36+x3_46+x3_56+x3_66
x37=x3_17+x3_27+x3_37+x3_47+x3_57+x3_67+x3_77
x38=x3_18+x3_28+x3_38+x3_48+x3_58+x3_68+x3_78+x3_88
x39=x3_19+x3_29+x3_39+x3_49+x3_59+x3_69+x3_79+x3_89+x3_99
x310=x3_110+x3_210+x3_310+x3_410+x3_510+x3_610+x3_710+x3_810+x3_910+x3_1010
x3=np.concatenate((x31,x32,x33,x34,x35,x36,x37,x38,x39,x310))
c2=x2/444*1000
c3=x3/444*1000
c_Feng2008=c2+c3
plt.plot(t1,c2,label='RISP')
plt.plot(t1,c3,label='9-OH')
plt.plot(t1,c_Feng2008,label='RISP+9-OH')
plt.legend()
plt.title('Feng2008')
plt.xlabel('t (h)')
plt.ylabel('c (ng/ml)')
plt.show()
此段代码过于冗长,有待优化。
图形如下:
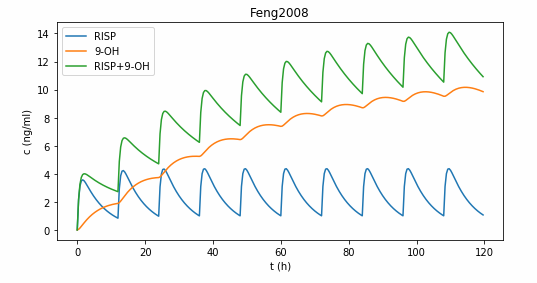
2.3 model2、3、4
model 1完成,下面画model 2、3、4,步骤几乎完全一样,只是模型稍有不同。model 2的模型和model 1完全一致;model 3中,利培酮多了一个周边室,是一个三室模型,所以方程更复杂一点;
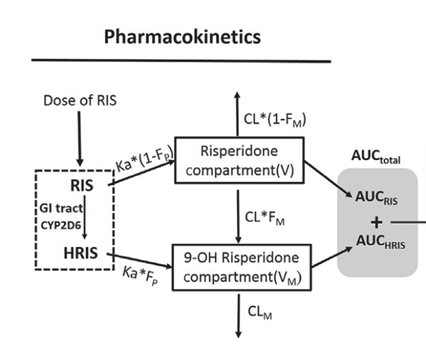
model4中吸收部位有一个向9-羟利培酮转化的过程。
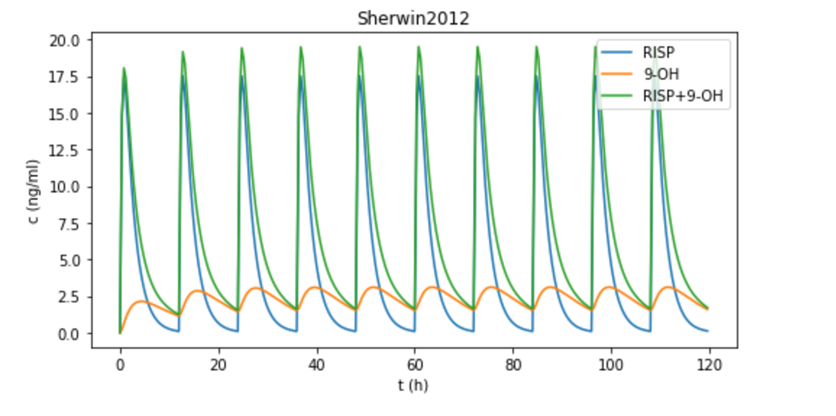
分别画出model2、3、4的多剂量给药图形如下:
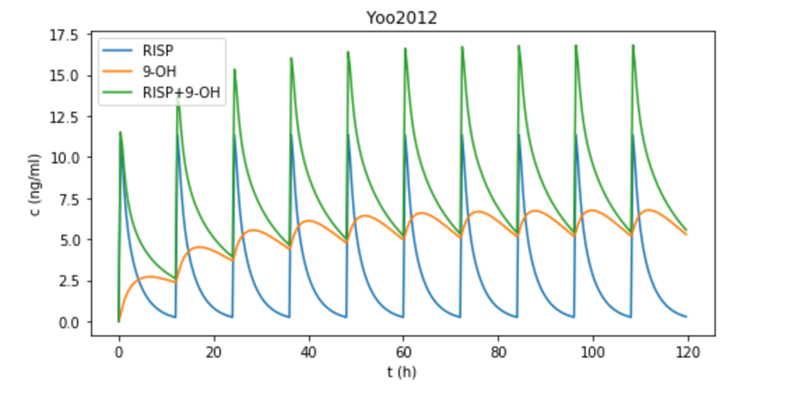
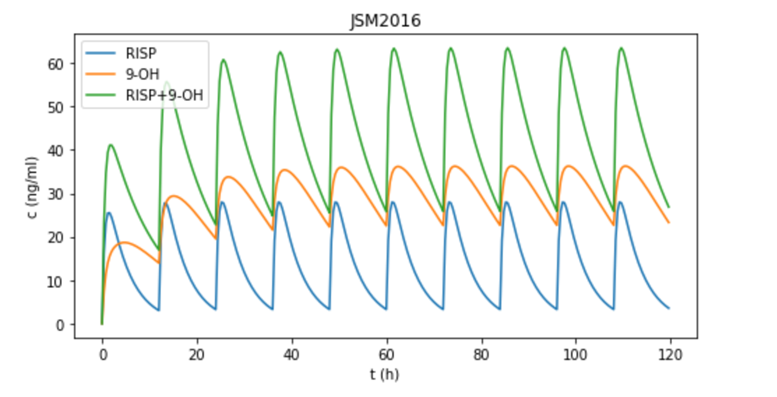
将4篇文献中的总浓度画在同一张图上,可以比较4个模型的稳态血药浓度范围: